Übungsaufgaben¶
Wenn Sie keine Note benötigen, können Sie die Übungsaufgaben (nicht die „Weiteren Aufgaben“) bis zum Dienstag der Folgewoche, 12 Uhr mittags, auf Papier oder per Email einreichen.
Beginnen Sie den Betreff der Email unbedingt mit „[BCM #Nummer] Ihr Name, Matrikelnummer“ inklusive der eckigen Klammern und des Hashtags, damit ich die Mails automatisiert sortieren kann.
Zum Beispiel: [BCM #4] Max Mustermann, 1234567
Die Übungsaufgaben werden in der folgenden Vorlesung kurz besprochen und müssen für den Abschluss mit Studienleistung bearbeitet werden. Es kommt dabei nicht primär auf die richtigen Lösungen an, sondern dass die Beschäftigung mit dem Thema erkennbar ist. (Eine richtige Lösung ist natürlich eine besonders gut erkennbare Beschäftigung mit dem Thema.)
#1 (VL am 18.04., Abgabe bis 25.04. 12 Uhr)¶
Warum spielt die OH-Gruppe von Tyrosin als deprotonierbare Gruppe eine Rolle, die der viel häufiger vorkommenden Aminosäuren Serin und Threonin aber nicht?
„Tris“ (Tris-(hydroxymethyl)-aminomethan) ist ein häufig verwendeter Puffer, der als „freie Base“ und als Hydrochlorid gehandelt wird. Was ist der Unterschied? Sind die Lösungen der reinen Substanzen basisch, neutral oder sauer?
In Phosphatpuffer lösen sie 22 mg/ml Rinderserumalbumin (bovine serum albumin, reife Form hat ein Mol.gew. von 66 kD). Wie hoch ist dann in der Lösung die Gesamtkonzentration titrierbarer, also (de)protonierbarer Gruppen des Proteins? (Link zum Uniprot-Eintrag)
Beweise, dass das Mischungskreuz funktioniert!
#2 (VL am 25.04., Abgabe bis 02.05. 12 Uhr)¶
Ordnen Sie die folgenden Methoden für den Pufferaustausch bei 10 ml einer Proteinlösung nach der steigender Effektivität, d.h. nach fallender Restkonzentration des „alten“ Puffers!
Dialyse gegen 1 l Puffer
Dialyse gegen 2x 0,5 l Puffer
6mal nacheinander: konzentrieren auf 2 ml und auffüllen auf 10 ml
3mal nacheinander: konzentrieren auf 0,5 ml und auffüllen auf 10 ml
Sie besitzen nach einer Plasmidpräparation 30 µl einer DNA-Lösung (das Plasmid ist 4,3 kb (Kilobasen) groß). 1 µl dieser Lösung wird 1:200 verdünnt und gemessen, die OD260 beträgt 0,43 Einheiten. Wie hoch ist die Stoffmengenkonzentration des Plasmids in der Stammlösung. Welche Gesamtmasse an DNA haben sie präpariert?
Lysozym aus Hühnereiweiß (MW 16,2 kD) hat die AS-Sequenz
MRSLLILVLCFLPLAALGKVFGRCELAAAMKRHGLDNYRG YSLGNWVCAAKFESNFNTQATNRNTDGSTDYGILQINSRW WCNDGRTPGSRNLCNIPCSALLSSDITASVNCAKKIVSDG NGMNAWVAWRNRCKGTDVQAWIRGCRLDie 1:20-Verdünnung einer Stammlösung zeigt bei 280 nm eine Absorption von 0,22 OD (1 cm Schichtdicke). Welche Konzentration hat die Stammlösung?
Warum können Sie die Proteinkonzentration in Fruchtsäften nicht zuverlässig mit dem BCA-Test messen?
#3 (VL am 02.05., Abgabe bis 09.05. 12 Uhr)¶
Zu welchem Gen gehört die folgende Nukleotidsequenz?
attggagctcgtaaggctggtaaagaactcgaacaattgagtaaaacgttgaaaattccaTipp: Durchsuchen Sie eine Gendatenbank!
Wenn bei der DNA-Synthese die korrekte Kupplungseffizienz pro Base 99% beträgt, wie lang kann dann die bei einer Synthese hergestellte DNA-Sequenz werden, wenn sie mit 10% korrektem Endprodukt noch zufrieden sind?
Bonus: Der häufigste Fehler bei der Synthese ist eine unvollständige Umsetzung: bei einem Schritt wird die Kupplung des Bausteins zufällig verpasst, in der nächsten Runde geht es aber normal weiter. Dadurch entstehen Sequenzen, in denen mittendrin eine Base fehlt. Angenommen, sie synthetisieren unter den o. g. Voraussetzungen eine DNA-Sequenz, die einhundert Basen lang sein soll. Welcher prozentuale Anteil der Reaktionsprodukte ist korrekt, welcher ist um eine Base, welcher um zwei Basen kürzer?
#4 (VL am 09.05., Abgabe bis 16.05. 12 Uhr)¶
DNA wird mit Restriktionsenzymen mit den folgenden Erkennungssequenzen verdaut.
SapI: 5‘-GCTCTTC-3‘
DraI: 5‘-TTTAAA-3‘
HhaI: 5‘-GCGC-3‘
Wie groß sind dann die erwarteten Bruchstücke durchschnittlich?
Beim Verdau eines Plasmids (eines ringförmigen DNA-Moleküls) mit den verschiedenen Restriktionsenzymen und anschließender Gelelektrophorese sehen Sie die folgenden Fragmente:
EcoRI: 1200 bp, 4800 bp
BamHI: 400 bp, 1900 bp, 3700 bp
EcoRI + BamHI (ein sogenannter Doppelverdau): 100 bp, 300 bp, 900 bp, 1000 bp, 3700 bp
Zeichnen Sie eine Plasmidkarte mit Schnittstellen, die dieses Muster ergeben! Ist das die einzige Möglichkeit?
#5 (VL am 16.05., Abgabe bis 23.05. 12 Uhr)¶
Wieviele (doppelsträngige) Kopien eines Template-Moleküls erhalten Sie nach 10 Zyklen einer „normalen“ PCR bzw. des QuikChange-Protokolls?
Können Sie PCR auch ausgehend von einem einzelsträngigen Templat durchführen?
Angenommen, Sie verwenden für das QuikChange-Protokoll ein Primerpaar, das nicht die identischen Bereiche in der Sequenz der zu mutierenden Stelle abdeckt (wie es korrekt wäre: Primer 1 + Primer 2), sondern zueinander leicht versetzt sind (Primer 1 mit Primer 2a, bzw. Primer 1 mit Primer 2b). Was für Fehler würden Sie dann erwarten?
Beispiel:
Primer 1 |------------------------> Template 5'-attggagctcgtaaggctggtaaagaactcgaacaattgagtaaaacgttgaaaattcca-3' Template 3'-taacctcgagcattccgaccatttcttgagcttgttaactcattttgcaacttttaaggt-5' Primer 2 <------------------------| Primer 2a <------------------------| Primer 2b <------------------------|Ein kommerzielles Kit für einen genetischen Fingerabdruck enthält Primer für 16 verschiedene Loci, für jeden Locus gibt es 15 unterscheidbare Allele, die alle mit gleicher Häufigkeit vorkommen. Wie groß ist dann näherungsweise die Wahrscheinlichkeit, dass zwei verschiedene Personen zufällig einen identischen genetischen Fingerabdruck mit diesem Kit produzieren? Berücksichtigen Sie, dass das Genom diploid ist. (Diese Annahmen sind alle stark vereinfachend.)
Wozu dient der Sequenzabschnitt zwischen den Positionen 2261 und 3118 auf dem Plasmid pET-20b(+)?
#6 (VL am 23.05., Abgabe bis 30.05. 12 Uhr)¶
Durch welche genetischen Eigenschaften unterscheidet sich der E.coli-Stamm „XL1 Blue“ von seinem Herkunftsstamm K12? Wählen sie drei Merkmale aus und beschreiben Sie die Funktion dieser genetischen Elemente mit ihren eigenen Worten.
Ein Gen wird in die mcs des Plasmids pET-51b(+) eingefügt und ein geeigneter Expressionsstamm (nennen Sie ein Beispiel) wird mit diesem Plasmid transformiert. Zu einer Flüssigkultur der resultierenden Zellen wird IPTG gegeben. Beschreiben sie mit ihren eigenen Worten die relevanten Prozesse, die daraufhin in den Zellen ablaufen!
Das lac-Repressor-Protein (das Genprodukt von lacI) hat zwei Sorten von Bindestellen, eine für den Induktor Allolactose, die andere für die Operator-DNA-Sequenz vor dem lac-Operon.
Was wären die physiologischen Auswirkungen für die E.coli-Zelle, wenn jeweils eine dieser Bindungsstellen bzw. beide gleichzeitig durch Mutationen nicht mehr zur Bindung des jeweiligen Liganden in der Lage wären?
Was passiert, wenn der lac-Operator so mutiert wird, dass der lac-Repressor ihn nicht mehr erkennt?
#7 (VL am 30.05., Abgabe bis 06.06. 12 Uhr)¶
Warum eignet sich die Silberfärbung nicht, wenn die Proteine hinterher noch durch analytische Verfahren wie z. B. Massenspektrometrie weiter untersucht werden sollen?
Erklären Sie folgende experimentelle Beobachtungen mit einer möglichst einfach Hypothese:
Eine Proteinprobe ergibt bei der isoelektrischen Fokussierung zwei eng benachbarte Banden
Dieselbe Proteinprobe ergibt im SDS-Gel genau eine Bande (innerhalb des Auflösungsvermögens der Methode)
Massenspektrometrie ergibt zwei Peaks, die Molekulargewichte der beiden Spezies unterscheiden sich um 80 Masseneinheiten.
Mitunter kann man die katalytischen Eigenschaften eines Proteins dazu ausnutzen, es in einem Elektrophorese-Gel zu identifizieren (wenn es z. B. Farbstoff produzieren kann). Warum funktioniert das aber normalerweise nicht bei der am häufigsten verwendeten SDS-PAGE?
Sie vergessen bei einem Westernblot die Behandlung der Membran mit dem aufgelösten Milchpulver (wie heißt dieser Schritt?). Was für ein Ergebnis erwarten Sie, nachdem Sie die Blotmembran mit dem primären und dem sekundären Antikörper behandelt haben?
#8 (VL am 06.06., Abgabe bis 13.06. 12 Uhr)¶
Ein Protein, das Sie isolieren möchten, stammt ursprünglich aus Thermus thermophilus. Sie haben das entsprechende Gen (1800 bp) kloniert und in E. coli auch erfolgreich nativ überexprimiert. Das Protein hat einen pI von 4 und ist bei pH 7 besonders stabil. Es bildet dann Homohexadecamere. Welche Reinigungsverfahren würden Sie auf jeden Fall ausprobieren?
Sie möchten die Thylakoidmembranen aus Spinat reinigen, weil sie im Praktikum damit die photosynthetische Sauerstoffproduktion messen wollen. Was wäre ein schlauer erster Reinigungsschritt, um die Proteine des Zytoplasmas möglichst bequem und vollständig von den Thylakoidmembranen abzutrennen?
Das Medium in einer Gelfiltrationssäule bestehe aus kleinen, kugelförmigen, porösen Partikeln gleicher Größe. Das Gelbett in der Säule hat einen Durchmesser von 10 mm und ist 30 cm lang. Ein Probe wird aufgetragen, dann wird mit Puffer eluiert.
Nach welchem Volumen durchgepumpten Puffers würden Sie ein Protein im Auslauf erwarten, dass für die Poren des Gels zu groß ist?
Nach welchem Volumen durchgepumpten Puffers würden Sie eine niedermolekulare Substanz im Auslauf erwarten?
#9 (VL am 13.06., Abgabe bis 20.06. 12 Uhr)¶
Carboanhydrase ist ein Enzym, das die Umwandlung von Kohlendioxid zu Kohlensäure katalysiert. Es spielt für viele biologische Prozesse eine Rolle und ist deshalb sehr gut untersucht. Eine Kristallstruktur hoher Auflösung finden Sie in der Proteinstrukturdatenbank unter der ID 3HS4.
Wie viele Lysin- und Cysteinreste enthält das Enzym?
Wie viele befinden sich davon exponiert auf der Oberfläche, so dass sie für Modifizierungsreagenzien zugänglich sind? (Zur Beantwortung ist ein 3D-Strukturvisualisierungstool hilfreich. Auf der PDB-Webseite ist eins integriert. Flexibler sind aber externe Programme wie ChimeraX oder PyMol .)
Angenommen, alle 20 Aminosäuren kommen mit gleicher Häufigkeit in Proteinen vor. (Das trifft bei weitem nicht zu, vereinfacht aber die Rechnung.) Wie viele Schnittstellen der folgenden Proteasen erwarten Sie dann im Durchschnitt für ein Protein, das aus 100 Aminosäuren besteht?
Chymotrypsin
Papain
TEV-Protease
Zeichnen Sie die chemische Struktur des Reaktionsproduktes von Pyrenbutansäuresuccinimidylester (siehe Vorlesungs-PDF) und einer Lysin-Seitenkette.
#10 (VL am 20.06., Abgabe bis 27.06. 12 Uhr)¶
Für Ihre Experimente möchten Sie verschiedene Proteine produzieren, die künstliche Aminosäuren an bestimmten Positionen enthalten. Schätzen Sie ein, wie schwierig es jeweils ist, die hier genannten Proteine herzustellen. Wie würden Sie in den Fällen, in denen die Aufgabe lösbar ist, die künstlichen Aminosäuren einführen?
ein Protein, das ein Azidohomoalanin und kein Methionin in der Sequenz enthält
ein Protein, das ein Azidohomoalanin und zwei weitere Methioninreste enthält
ein Protein, das zwei Azidohomoalanine und kein Methionin enthält
ein Protein, das para-Cyanophenylalanin enthält
ein Protein, das para-Cyanophenylalanin und Azidohomoalanin und kein Methionin enthält
ein Protein, das para-Cyanophenylalanin, Azidohomoalanin und Methionin enthält
ein Protein, das para-Cyanophenylalanin und para-Azidophenylalanin enthält
Warum können Sie als 13C-Quelle für die Isotopenmarkierung in E. coli nicht 13C-Natriumhydrogencarbonat verwenden, das viel billiger wäre als 13C-Glucose oder 13C-Glycerol?
#11 (VL am 27.06., Abgabe bis 04.07. 12 Uhr)¶
Cyclophilin A ist eine Peptidyl-Prolyl-cis/trans-Isomerase. Das Enzym katalysiert eine der einfachsten denkbaren chemischen „Reaktionen“: die Rotation um die N-terminale Peptidbindung eines Prolin-Restes in einer Polypeptidkette. Obwohl sich Substrat und Produkt der Reaktion nur durch die Konformation (cis oder trans) einer einzigen Bindung unterscheiden, lässt sich die Aktivität des Enzyms mit einem sehr eleganten Trick messen.
Wie funktioniert dieser Aktivitätstest? Lesen Sie dazu das (kurze) Paper von Fischer et al. und beschreiben Sie den Test mit eigenen Worten.
#12 (VL am 04.07., Abgabe bis 11.07. 12 Uhr)¶
Ein Protein P bindet einen Liganden L, es entsteht der Komplex PL (bestehend aus je einem Molekül Protein und Ligand). Diese Gleichgewichtsreaktion hat die Dissoziationskonstante
, die sie bestimmen möchten. Dazu verwenden sie eine Fluoreszenzmessung bei einer geeigneten Wellenlänge. P allein hat eine spezifische Fluoreszenz von
, der Komplex hat eine spezifische Fluoreszenz von
. Das sichtbare Fluoreszenzsignal ist dann proportional zur Konzentration der jeweiligen Spezies, d. h. beispielsweise für das Protein:
. Der Ligand allein fluoresziert nicht. Zu einer Lösung von P geben sie schrittweise immer mehr Ligand L hinzu und messen dabei die Fluoreszenz
der Lösung in Abhängigkeit von der Ligandenkonzentration. Sie können vereinfachend annehmen, dass sich das Volumen der Lösung durch die Zugabe von L nicht ändert.
Leiten Sie eine Gleichung für die Fluoreszenz der Lösung in Abhängigkeit von der Ligandenkonzentration, der Proteinkonzentration und der Dissoziationskonstante her, die sie dann benutzen könnten, um
durch einen Fit der Messkurve zu ermitteln!
Achtung
Am 11.07. findet keine Vorlesung mehr statt. Allen Teilnehmern und Teilnehmerinnen, die bis zum 11.07. Lösungen zu mindestens 9 Übungszetteln eingereicht haben, bescheinige ich die entsprechende Studienleistung. Bitte teilen Sie mir mit, wenn diese Meldung nicht an das Prüfungsamt Physik gehen soll.
Bis zum 11.07. ist auch noch die Evaluation der Veranstaltung möglich.
Lösung zu #12¶
Mit den Gleichgewichtskonzentrationen ![[P], [L], [PL]](_images/math/1c55195b131ff38dc3285a6ea596c2e546708a52.png) von Protein, Ligand und Komplex gilt per Definition der Dissoziationskonstanten:
von Protein, Ligand und Komplex gilt per Definition der Dissoziationskonstanten:
![K_D = \frac{[P][L]}{[PL]}](_images/math/dc6d1c79375330420f68d2cf843d57fa9412e224.png)
Die Konzentrationen im Gleichgewicht sind jedoch nicht bekannt, nur die Ausgangskonzentrationen ![[P]_0, [L]_0](_images/math/684cfed5ad313fabdb94f3f32f78902069486f80.png) von Protein und Ligand. Wir wissen nur, dass sich die Ausgangskonzentrationen dieser beiden im Gleichgewicht um die gebildete Menge Komplex verringert haben müssen. Anders gesagt: im Gleichgewicht ist nur noch so viel frei, wie nicht im Komplex gebunden ist. Eingesetzt ergibt sich damit:
von Protein und Ligand. Wir wissen nur, dass sich die Ausgangskonzentrationen dieser beiden im Gleichgewicht um die gebildete Menge Komplex verringert haben müssen. Anders gesagt: im Gleichgewicht ist nur noch so viel frei, wie nicht im Komplex gebunden ist. Eingesetzt ergibt sich damit:
![K_D = \frac{([P]_0-[PL])([L]_0-[PL])}{[PL]}](_images/math/4517900effa02947a781eff7a11b647cca874db4.png)
Diese quadratische Gleichung in ![[PL]](_images/math/8200f39e3c92f25d7ae074d3486aceafa3cd9129.png) hat nur eine chemisch sinnvolle Lösung (warum ist die andere nicht relevant?):
hat nur eine chemisch sinnvolle Lösung (warum ist die andere nicht relevant?):
![[PL] = \frac{[P]_0+[L]_0+K_D}{2} - \sqrt{\left (\frac{[P]_0+[L]_0+K_D}{2}\right )^2-P_0 L_0}](_images/math/e3358d2d70e46755b09793018af241ee354654ec.png)
Die Gesamtfluoreszenz 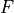 ergibt sich wie folgt:
ergibt sich wie folgt:
![F = F_P \cdot [P] + F_{PL} \cdot [PL]
F = F_P \cdot ([P]_0 - [PL]) + F_{PL} \cdot [PL]
F = F_P \cdot [P]_0 + (F_{PL}-F_{P})\cdot [PL]](_images/math/b0f368e161cc737ed3892367e807d13d998f4870.png)
Man kann nun noch den obigen Ausdruck einsetzen und erhält schließlich:
![F = F_P \cdot [P]_0 + (F_{PL}-F_{P})\cdot \left (\frac{[P]_0+[L]_0+K_D}{2} - \sqrt{\left (\frac{[P]_0+[L]_0+K_D}{2}\right )^2-P_0 L_0}\right )](_images/math/0893eedf5b0fbded4042e0c134346b17dda431e2.png)
Diese Gleichung enthält jetzt bis auf 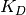 nur noch bekannte Größen und kann an die Messkurve gefittet werden.
nur noch bekannte Größen und kann an die Messkurve gefittet werden.
Hier ist ein interaktiver Beispielplot bei Desmos. 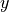 ist darin das Fluoreszenzsignal,
ist darin das Fluoreszenzsignal, 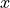 ist die Ligandenkonzentration und
ist die Ligandenkonzentration und 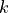 der Logarithmus von
der Logarithmus von 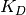 (damit man einfach mehrere Größenordnungen abfahren kann.)
(damit man einfach mehrere Größenordnungen abfahren kann.)